Em mais um tópico da coluna ‘Drops de Genômica’, o oncologista André Murad (foto) explica o “splicing” (emenda) alternativo, processo no qual um único gene pode gerar múltiplas moléculas de mRNA, selecionando diferentes combinações de éxons durante o processamento do RNA. Confira.
Por André Marcio Murad*
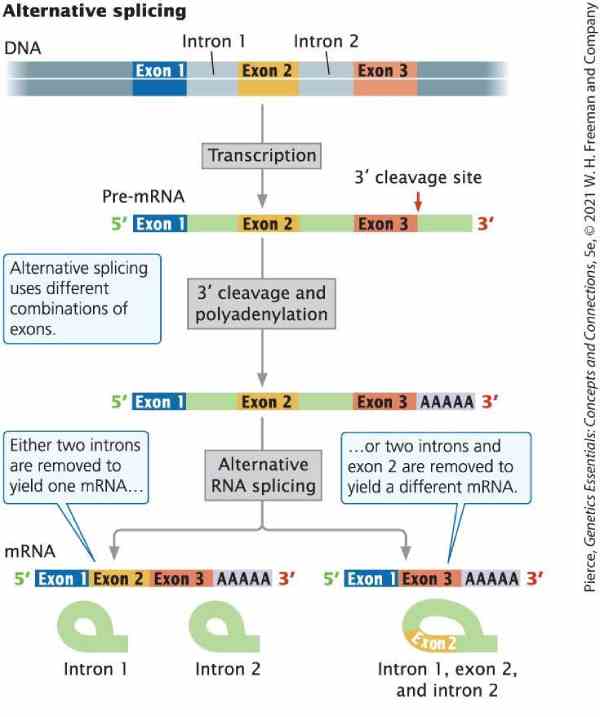
O DNA contém três éxons (1, 2 e 3) separados por íntrons. Após a transcrição, uma molécula de pré-mRNA é produzida, incluindo todos os éxons e íntrons.
Durante o processamento do RNA, o pré-mRNA sofre clivagem na extremidade 3′ e poliadenilação, adicionando uma cauda poli-A para estabilizar o transcrito. O “splicing” alternativo determina, então, quais éxons serão retidos.
Em uma via, ambos os íntrons são removidos, produzindo um mRNA que contém o éxon 1, o éxon 2 e o éxon 3.
Em outra via, o íntron 1, o éxon 2 e o íntron 3 são removidos simultaneamente, resultando em um mRNA que une o éxon 1 diretamente ao éxon 3.
Esse mecanismo permite que um único gene produza proteínas diferentes, dependendo de quais éxons estão incluídos no mRNA final.
A desregulação do “splicing” alternativo está fortemente associada ao câncer. Na carcinogênese, esse processo pode ser alterado por mutações em fatores de “splicing”, levando a variantes de “splicing” anormais que promovem o desenvolvimento tumoral ao afetar a proliferação celular, a resistência à apoptose, a invasão, a metástase e a evasão imunológica. Isso torna o “splicing” alternativo aberrante um alvo potencial para novos diagnósticos e terapias contra o câncer.
*André Murad é diretor científico do Grupo Brasileiro de Oncologia de Precisão (GBOP), diretor clínico da Personal - Oncologia de Precisão e Personalizada, professor adjunto coordenador da Disciplina de Oncologia da Faculdade de Medicina da UFMG, e oncologista e oncogeneticista da Clínica OncoLavras





